Une étude dirigée par des chercheurs de l’Institut Pasteur de Paris rapporte les premières données épidémiologiques, virologiques et biologiques concernant le variant XD du SARS-CoV-2, issu de la recombinaison entre les variants Delta et Omicron. Ces travaux, supervisés par Étienne Simon-Lorière, responsable du groupe ‘Génomique évolutive des virus à ARN’ à l’Institut Pasteur, ont été publiés le 4 avril 2022 sur la plateforme de prépublication Research Square.
Recombinaison génétique
Les coronavirus ont une grande capacité à se recombiner, autrement dit à échanger entre eux des séquences de leur matériel génétique, avec pour conséquence de générer des virus dont l’ARN comporte des régions provenant des deux virus parentaux. Lorsqu’une même cellule est infectée par deux variants du SARS-CoV-2 génétiquement distincts, la co-infection peut aboutir à la production d’un virus hybride ou recombinant, du fait d’un phénomène de recombinaison génétique.
L’ARN du coronavirus recombinant est classiquement le résultat d’un processus fort complexe (template switching mechanism) qui a lieu lors de la réplication du virus au sein de la cellule infectée*. Il arrive que l’enzyme ARN polymérase saute d’une molécule d’ARN (donneuse) à une autre (accepteuse), ce qui aboutit à la production d’une molécule d’ARN composite provenant de deux génomes parentaux. L’ARN qui en résulte renferme alors des portions de matériel génétique de chacun des deux virus parentaux.
Des virus SARS-CoV-2 recombinants ont été décrits dans la littérature scientifique durant la pandémie Covid-19. Récemment, plusieurs variants issus de la recombinaison entre les variants Delta et le sous-lignage BA.1 d’Omicron ont été identifiés, de même que des virus recombinants résultant d’une recombinaison entre les sous-lignages BA.1 et BA.2 d’Omicron.
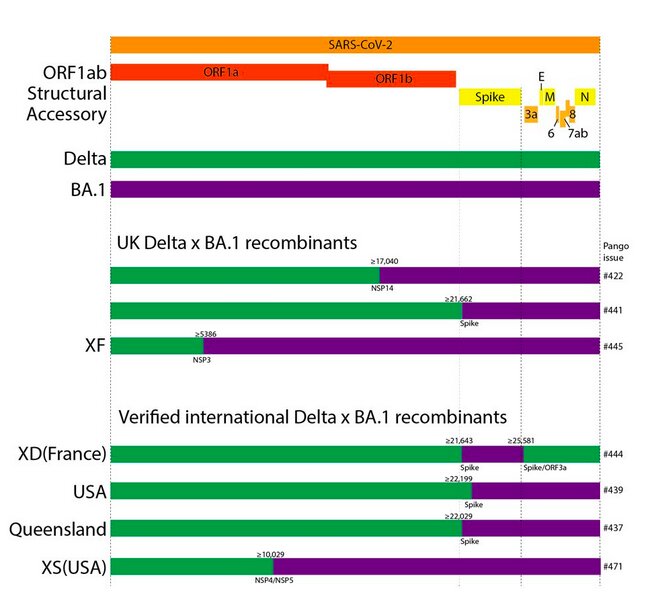
Début 2022, plusieurs laboratoires en France (consortium EMERGEN) et en Europe, impliqués dans la surveillance génomique, ont rapporté des séquences SARS-CoV-2 particulières. Des analyses plus poussées ont rapidement montré que ces génomes viraux renfermaient la signature mutationnelle caractéristique du sous-lignage Omicron mais seulement sur une partie de l’ARN viral, ce qui plaide fortement en faveur de l’hypothèse que ces virus résultent d’une recombinaison génétique.
Un examen approfondi des données brutes et du séquençage du génome d’un isolat viral a permis d’exclure la possibilité que l’on puisse être en présence de deux génomes différents ou d’une contamination de laboratoire par un second virus. Le virus recombinant en question a été dénommé XD et classé le 17 mars comme variant sous surveillance par le Centre européen de contrôle et de prévention des maladies (ECDC, European Centre for Disease Prevention and Control).
Les analyses moléculaires ont permis d’identifier dans l’ARN viral deux points de cassure (breakpoints), témoins d’une recombinaison des deux génomes parentaux à leur niveau. Dans le variant XD, l’un de ces points de cassure se situe au début de la séquence codant la protéine spike, l’autre au sein d’une région dénommée ORF3.
Données épidémiologiques
Le premier génome de ce nouveau variant XD a été identifié dans le nord de la France à l’occasion d’une surveillance génomique de routine lors d’une enquête « Flash ».
En date du 18 mars, ce sous-lignage XD a été détecté dans plusieurs régions en France, mais également au Danemark, aux Pays-Bas, en Belgique et en Allemagne. Dans la majorité des cas, ce virus recombinant a été identifié chez des individus qui n’avaient pas voyagé. L’un d’eux s’était cependant rendu en Italie, ce qui est en faveur d’une importante circulation du variant XD.
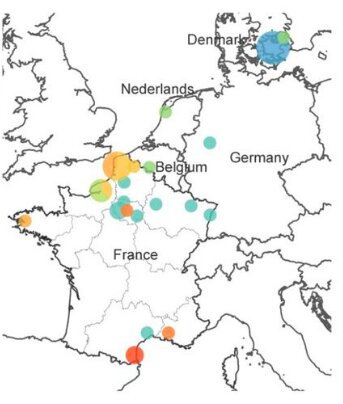
Selon Santé publique France, au 28 mars, 63 séquences caractéristiques du variant XD avaient été détectées dans notre pays. Ces séquences correspondent à des cas provenant de plusieurs régions de France métropolitaine, le variant XD représentant moins de 0,1% des séquences génomiques interprétables.
Données cliniques
Si la majorité des cas (54 %) avait bénéficié d’une primo-vaccination complète (deux doses), dont 24 % d’une première dose de rappel, seuls trois cas (6 %) avaient présenté une infection antérieure par le SARS-CoV-2, ce qui est plus faible qu’avec Omicron (14 % des 468 cas analysés entre novembre 2021 et janvier 2022).
Les chercheurs indiquent que l’âge médian des patients infectés par le variant XD est de 35 ans, similaire à ce que l’on observe avec Omicron. On dénombre cependant un plus grand nombre de cas d’infection par XD chez les moins de 20 ans (25,7 %) en comparaison avec Omicron (11,3 %). Aucun cas d’infection par XD n’a été observé chez les personnes âgées de plus de 70 ans.
En France, tous les cas d’infection par le variant XD étaient symptomatiques. La plupart des symptômes sont des maux de tête (61 %), de l’asthénie/fatigue (58 %), de la toux (44 %), de la fièvre (39 %), des douleurs musculaires (33 %). En comparaison avec Omicron, la proportion de cas ayant rapporté une perte de goût (agueusie) ou d’odorat (anosmie) était plus importante parmi les individus infectés par le variant XD. Deux patients ont été hospitalisés, mais aucun n’a dû être admis en réanimation.
Données génétiques
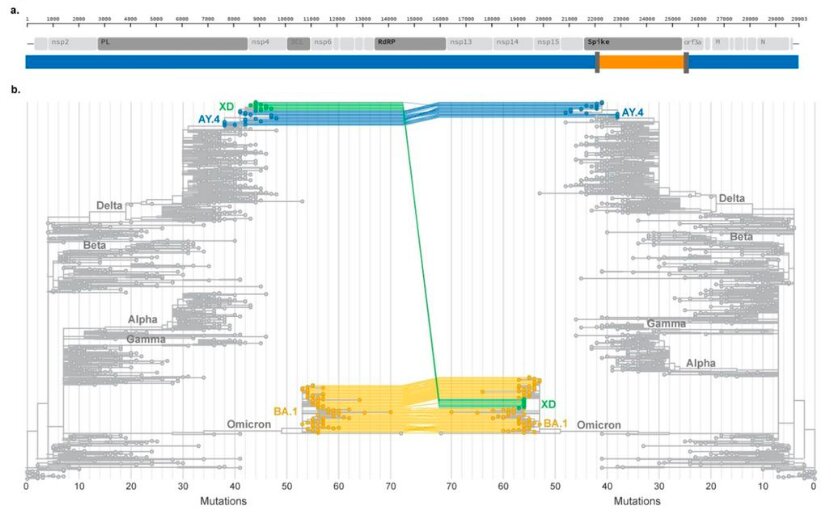
Concernant la caractérisation de XD sur le plan génétique, il ressort des analyses que la partie du génome de XD provenant du variant Delta correspond à un petit groupe de séquences correspondant au sous-lignage AY.4. Les séquences AY.4 apparentées les plus proches ont été identifiées en France, en Allemagne et en Suède. Tout indique que le variant recombinant XD forme un groupe bien à part, partageant un ancêtre commun unique (« cluster monophylétique »).
La région du génome du virus XD provenant du variant Delta correspond à 100 % à celle du virus AY.4 qui circulait en Ile-de-France fin décembre 2021. Quant à la région de XD issue du variant Omicron, elle est également 100 % identique à celle du sous-lignage BA.1 Omicron, précisent les auteurs. Ces résultats leur ont permis de rapidement caractériser XD en tant que virus recombinant issu de variants parentaux génétiquement distincts.
Échappement immunitaire
Les chercheurs ont ensuite analysé la capacité de neutralisation du virus XD par les anticorps produits par la vaccination. Le virus recombinant XD renferme l’intégralité du domaine de liaison du récepteur (RBD, receptor binding domain) du variant BA.1 Omicron. On rappelle que le RBD est la région de la protéine spike qui se lie au récepteur ACE2 sur les cellules que le virus infecte.
Les tests de neutralisation par anticorps ont utilisé des pseudovirus, c’est-à-dire d’autres virus que le SARS-CoV-2, artificiellement porteurs à la surface de la protéine spike du variant XD ou de celle de la souche ancestrale, du variant Delta (AY.4) ou encore d’Omicron (BA.1). Les sérums ayant servi à ces tests de neutralisation provenaient de sujets infectés et vaccinés avec le vaccin Pfizer, vaccinés avec deux doses du vaccin Pfizer ou AstraZeneca ou chacun de ces deux vaccins, ou ayant reçu un rappel avec le vaccin Moderna. Les prélèvements sanguins avaient été effectués un à six mois après la dernière injection vaccinale.
Il s’avère que XD possède des propriétés d’échappement immunitaire similaires à celle du sous-lignage BA.1 Omicron. Le variant recombinant XD, de même que BA.1 Omicron, a été très peu neutralisé par le sérum de patients ayant reçu deux doses de vaccin.
Les titres (quantités) en anticorps étaient beaucoup plus élevés un mois après la troisième dose. La capacité de neutralisation de XD (et du sous-variant BA.1 d’Omicron) était cependant 8 à 10 fois inférieure à celle observée avec la souche ancestrale. De même, les individus infectés n’ayant qu’une seule dose de vaccin ont présenté une capacité de neutralisation de BA.1 (Omicron) et XD respectivement 3,4 et 3,7 fois inférieure par rapport à la souche originelle (différence non significative).
Les chercheurs ont évalué la capacité d’anticorps monoclonaux thérapeutiques dirigés contre la protéine spike à neutraliser le variant XD. Il s’avère que le tixagévimab ne le neutralise pas. La capacité de neutralisation de l’anticorps cilgavimab est réduite de 13,5 fois.
Evusheld® (association des anticorps monoclonaux tixagévimab et cilgavimab) a un pouvoir neutralisant fortement réduit (de 37,9 fois contre XD, de 36,7 fois contre le sous-lignage BA.1 d’Omicron), alors que ce cocktail d’anticorps neutralise efficacement Delta (AY.4). La baisse du pouvoir neutralisant de l’anticorps sotrovimab est plus prononcée vis-à-vis de XD que contre le sous-variant BA.1 d’Omicron (réductions respectivement de 5,1 et 2,6 fois).
L’ensemble de ces résultats montre donc que le virus recombinant XD possède, à l’instar d’Omicron (BA.1), des capacités d’échappement immunitaire en termes d’efficacité vaccinale et de neutralisation par les anticorps monoclonaux thérapeutiques.
Réplication in vivo dans un modèle animal
La dynamique de la réplication du variant XD a également été évaluée in vivo dans un modèle animal de l’infection par le SARS-CoV-2, en l’occurrence des souris transgéniques exprimant le récepteur humain ACE2. Ces rongeurs ont été infectés soit par le virus recombinant XD, soit par chacun des deux variants parentaux de XD (Delta et Omicron). Les chercheurs ont ensuite évalué, trois jours après l’infection, la charge virale dans le tissu pulmonaire et les fosses nasales (cornets nasaux).
Les taux d’ARN viral dans les poumons des souris infectées par XD et le sous-variant BA.1 d’Omicron étaient comparables, à savoir 3,7 fois inférieurs à ceux observés chez les rongeurs infectés par Delta (AY.4).
En revanche, la charge virale dans les cornets nasaux des souris infectées par XD était 8,5 inférieure à celle observée chez les souris infectées par le sous-variant BA.1 d’Omicron.
Alors que les souris infectées par Delta (AY.4) présentaient une maladie associée à une perte de poids, celles infectées par le virus XD n’ont pas perdu de poids dans les cinq premiers jours post-infection mais elles ont toutes dépéri durant les trois jours suivants. Toutes sont mortes ou ont dû être euthanasiées. Les rongeurs infectés par Omicron (BA.1) n’ont en revanche pas perdu de poids et aucun n’est mort.
Ces résultats indiquent donc que le virus recombinant XD est associé, chez ces souris transgéniques pour le récepteur humain ACE2, à une pathologie différente de celle observée chez celles infectées par le sous-variant BA.1 d’Omicron. Il reste à déterminer quels déterminants moléculaires dans XD sous-tendent la sévérité de la maladie clinique chez les rongeurs en cas d’infection par ce variant recombinant et déterminer ce qu’il en est chez l’homme.
Enfin, les chercheurs ont évalué la capacité de réplication (fitness) du virus XD en comparaison avec le sous-lignage BA.1 d’Omicron, qui représentait le variant SARS-CoV-2 dominant en France et dans la plupart des pays européens au moment où XD a été identifié pour la première fois. Plutôt que d’infecter différents lots de souris avec Omicron et XD, les chercheurs ont inoculé à ces rongeurs des mélanges de ces deux virus dans des proportions différentes (rapports respectifs de 1/1, 1/9, 9/1) afin de savoir quel variant prenait le dessus sur l’autre trois jours après l’infection dans les poumons et dans les fosses nasales.
Capacité accrue de réplication dans les voies aériennes supérieures
Alors que le sous-lignage BA.1 d’Omicron dominait constamment XD dans les poumons, les chercheurs ont observé une situation inverse au niveau des cornets nasaux. Ces deux variants présentent donc un avantage pour la réplication différent selon le site anatomique.
Sachant que ces deux variants partagent la même séquence codant le RBD de la protéine spike, cette sous-région du génome viral n’est pas responsable de la grande capacité de XD à se répliquer dans les voies aériennes supérieures. Il reste à savoir si d’autres régions de la protéine spike, notamment le domaine N-terminal qui, lui, provient du variant Delta (AY.4), voire d’autres régions du génome, sous-tendent les différences observées.
Indispensable surveillance génomique
À ce stade des recherches, on ignore le pouvoir pathogène du variant XD chez l’homme de même que sa capacité de transmission, en particulier au sein d’une population déjà exposée à une infection naturelle ou qui a été vaccinée. Des études supplémentaires sont donc nécessaires pour déterminer plus précisément les caractéristiques épidémiologiques et les propriétés biologiques de ce variant recombinant.
Du fait de l’intense circulation du SARS-CoV-2 dans la population générale, qui contribue à augmenter les opportunités de recombinaison entre variants génétiquement distincts, il est « crucial » de continuer la surveillance génomique à l’échelle mondiale et de rapidement évaluer les caractéristiques biologiques d’éventuels variants préoccupants émergents, concluent les auteurs.
Marc Gozlan (Suivez-moi sur Twitter, Facebook, LinkedIn, et sur mon nouveau blog ‘Le diabète dans tous états’, consacré aux mille et une facettes du diabète. Déjà quatre billets)
* La recombinaison intervient lors de l’étape de réplication lors de laquelle des petits fragments d’ARN sont répliqués (ARN sous-génomiques). La production d’ARN sous-génomiques implique un processus de transcription discontinue : l’ARN génomique (+) est répliqué en ARN (-), qui sera à son tour transcrit de manière discontinue en brins de différentes tailles. Ce brin d’ARN (-) sert ensuite lui-même de brin matrice pour la synthèse de brins.
Pour en savoir plus :
Simon-Lorière E, Montagutelli X, Lemoine F. Rapid characterization of a Delta-Omicron SARS-CoV-2 recombinant detected in Europe. Research Square. Posted 04 Apr, 2022. doi: 10.21203/rs.3.rs-1502293/v1
Combes P, Bisseux M, Bal A, et al. Evidence of co-infection during Delta and Omicron variants of concern co-circulation, weeks 49-2021 to 02-2022, France. medRxiv. Posted March 03, 2022. doi: 10.1101/2022.03.02.22271694
Francisco Junior RDS, de Almeida LGP, et al. Emergence of Within-Host SARS-CoV-2 Recombinant Genome After Coinfection by Gamma and Delta Variants: A Case Report. Front Public Health. 2022 Feb 22;10:849978. doi: 10.3389/fpubh.2022.849978
Wertheim JO, Wang JC, Leelawong M, et al. Capturing intrahost recombination of SARS-CoV-2 during superinfection with Alpha and Epsilon variants in New York City. medRxiv. Posted January 21, 2022. doi: 10.1101/2022.01.18.22269300
Amoutzias GD, Nikolaidis M, Tryfonopoulou E, et al. The Remarkable Evolutionary Plasticity of Coronaviruses by Mutation and Recombination: Insights for the COVID-19 Pandemic and the Future Evolutionary Paths of SARS-CoV-2. Viruses. 2022 Jan 2;14(1):78. doi: 10.3390/v14010078
VanInsberghe D, Neish AS, Lowen AC, Koelle, K. Recombinant SARS-CoV-2 genomes circulated at low levels over the first year of the pandemic. Virus Evolution. 2021 Dec;7(2):veab059. doi: 10.1093/ve/veab059
Gutierrez B, Castelán-Sánchez HG, Candido DDS, et al. Emergence and widespread circulation of a recombinant SARS-CoV-2 lineage in North America. medRxiv. Posted November 21, 2021. doi:10.1101/2021.11.19.21266601
Jackson B, Boni MF, Bull MJ, et al. Generation and transmission of interlineage recombinants in the SARS-CoV-2 pandemic. Cell. 2021 Sep 30;184(20):5179-5188.e8. doi: 10.1016/j.cell.2021.08.014
Boni MF, Lemey P, Jiang X, et al. Evolutionary origins of the SARS-CoV-2 sarbecovirus lineage responsible for the COVID-19 pandemic. Nat Microbiol. 2020 Nov;5(11):1408-1417. doi: 10.1038/s41564-020-0771-4
Simon-Loriere E, Holmes EC. Why do RNA viruses recombine? Nat Rev Microbiol. 2011 Jul 4;9(8):617-26. doi: 10.1038/nrmicro2614
Sur le web :
COVID-19 : point épidémiologique du 31 mars 2022 (Santé publique France). Mis à jour le 1 avril 2022
Recombinant variants detected in the UK data (COG-UK Mutation Explorer)
Pourquoi une telle différence de la capacité de neutralisation de XD entre vaccinés 3 doses (8-10 fois moins) et infectés+1 dose (3-4 fois moins)?
On ne « démarre » pas avec le même taux de base dans les deux cas. Il est possible que cette différence apparente soit également due à une diversification du type d’anticorps observés dans la situation ‘infection puis vaccination’ par rapport à la vaccination avec trois doses. Les tests de neutralisation ne détecteraient donc peut-être pas tout à fait la même chose. Pour débrouiller ces résultats préliminaires et parcellaires, il importe surtout de disposer de données reposant sur de véritables virus et non sur des pseudovirus. C’est probablement l’objet des recherches en cours.
Bonjour, merci de votre retour. J’ai l’impression que beaucoup de tests de caractérisation de la réponse immunitaire se focalisent sur les anticorps, et y a-t-il des recherches (et résultats) sur les autres mécanismes de notre système immunitaire, mais aussi sur des différences entre vaccination (types, doses) et infection (et toutes le panel entre les deux)?
L’immunité cellulaire, beaucoup plus laborieuse à explorer que l’immunité humorale, fait également l’objet d’intenses recherches, notamment chez les vaccinés.
Quelques références récentes :
Analysis of mRNA vaccination-elicited RBD-specific memory B cells reveals strong but incomplete immune escape of the SARS-CoV-2 Omicron variant
Dynamics of humoral and T-cell immunity after three BNT162b2 vaccinations in adults older than 80 years
Merci pour les liens et bonne continuation pour ce blog !